星状凸多面体のアイデアを用いた、効率的なセグメンテーションモデル : STAR-DIST3Dが登場
論文 : Star-convex Polyhedra for 3D Object Detection and Segmentation in Microscopy
written by Martin Weigert, Uwe Schmidt, Robert Haase, Ko Sugawara, Gene Myers
Submitted on 9 Aug 2019
Subjects: Computer Vision and Pattern Recognition (cs.CV)
さて、AIの実用化でホットな話題として、「AI×医学」があります。医学とテクノロジーの融合である”Med-tech”の一分野として、ビジネス方面からも熱い注目を受けていますね。
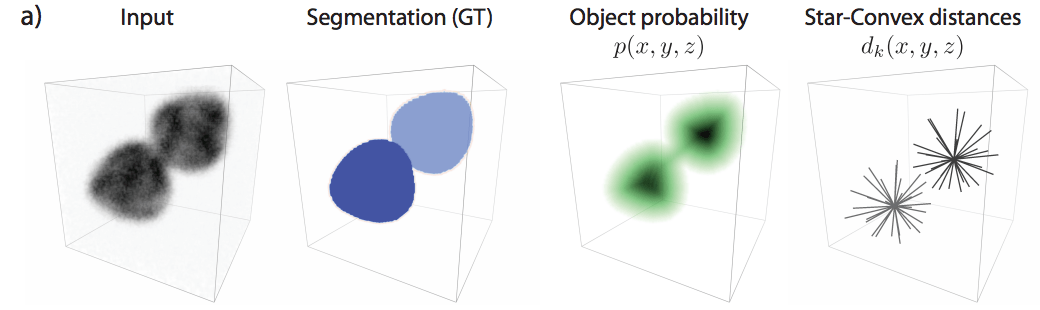
医学の実験は、その”細胞”に注目することから始まり、さらにその中心である”核”の研究に到達します。それら細胞やその核は、その性質などや場所によって様々な種類に分けられます。
しかしながら、その種類に対して”色”や”形”は非常に単一的であり、実際に区別をつけたり、異常を発見するには相当の訓練が必要でしょう。(余談ですが、白血球が白くないと初めて知った時、なぜか当時小学生の僕はショックを受けたのを非常に覚えています・・・!)
今回紹介するモデル「STAR-DIST3D」は”細胞核”のデータセットに対して、3次元におけるセグメンテーションを行います。細胞の成長度合いごとの色合いを元に、各細胞の色付けを行うことが可能になります。
従来の細胞核データセットにおけるセグメンテーションは、2D表現が限界でした。そこに、3Dへのモデル拡張と星状凸多面体表現の適用を試みます。ただ2次元向けに作成されたモデルを3次元に拡張しようとすると計算量が爆発してしまうため、工夫が必要となります。
モデル概要
細胞核の星状凸多面体表現における効率的なパラメータを発見し、蛍光顕微鏡のデータセットにその表現方法を適用します。それに加えて、2つの星状凸多面体の交差領域を、NMSを用いることで効率的に計算し、3D表現の獲得を目指します。
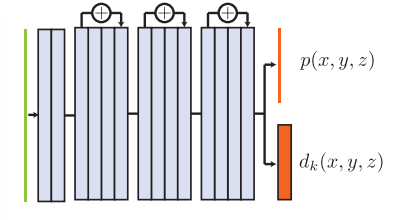
主な流れは、ResNetと同様になっており、dは半径方向距離(radial distance)を、pはユークリッド距離における物体の存在確率を確率分布で表しています。入力画像を元に、まずセグメンテーションを行い、そこからdとpの計算を学習します。
・NMS (Non-maximum suppression)の導入
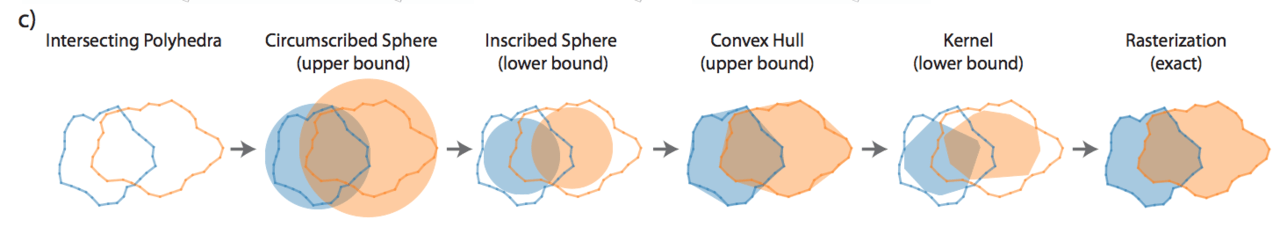
入力された画像において物体検出を行なった際、2つの物体が占める領域が重複した部分が出てしまった場合、それらを目的に応じて適切に処理する必要があります。この問題を”効率的に”計算するのは簡単な問題ではありません。
「STAR-DIST3D」では、入力画像内の2つの交差した領域における正確な処理を行うため、NMS(Non-maximum suppression)を導入しています。
上画像は、NMSにおける計算の流れを図示したものになります。重なっている部分の体積を様々な手法により計算し、正解データとの誤差を計算しながら学習を行います。
はじめに、2つの重複を含む物体をそれぞれ認識し、バウンディングボックス (外枠)を生成します。次に、検出された2つの物体から外接円と内接円を計算し、それと同時に凸包(与えられた点をすべて包含する最小の凸多角形(凸多面体))とカーネルを計算します。もし、この作業の繰り返しにより生成されたバウンディングボックスが、正解データと差異が少なくなるパラメータを学習させていきます。
この凸多面体の計算アイデアの導入により、本モデルはU-NETと比較し、計算所要時間を削減しながら制度を向上させることに成功しています。
より、星状凸多面体表現における2次元セグメンテーションについて知りたい方は、以下の論文を参考にしていただけると良いでしょう。 参考論文 : Cell Detection with Star-convex Polygons
実験結果
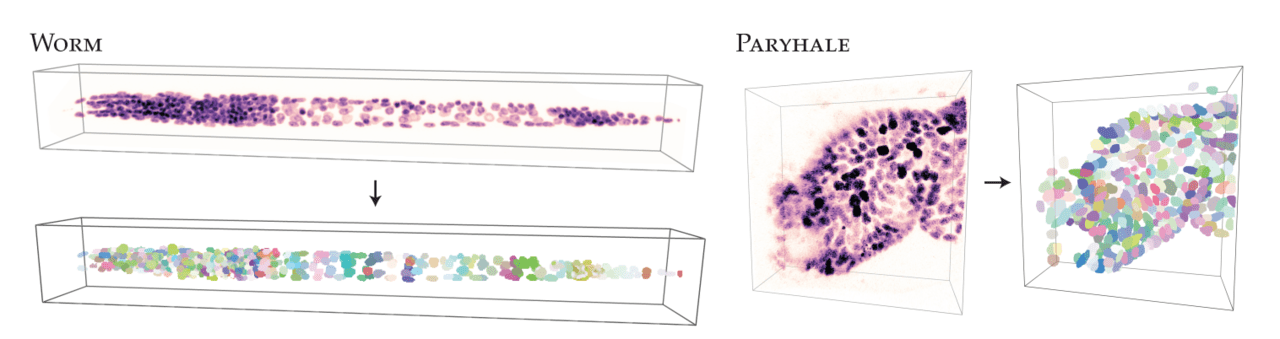
核細胞のセグメンテーション実験には、「WORM」と「PARYHALE」の2種類のデータセットを用います。両方とも3次元空間におけるデータセットであり、白〜紫色で細胞核を成長段階ごとに表した生のデータと、それらをラベルごとに色分けした正解データの2つからなっています。
PARYHALEは異方的(回転によってその性質が変化する)なデータであり、WORMは等方的(その性質が回転に依存しない)データです。
(i)比較実験
「STAR-DIST3D」により2次元の細胞核セグメンテーションの実験を行い、既存モデルとの比較を行いました。
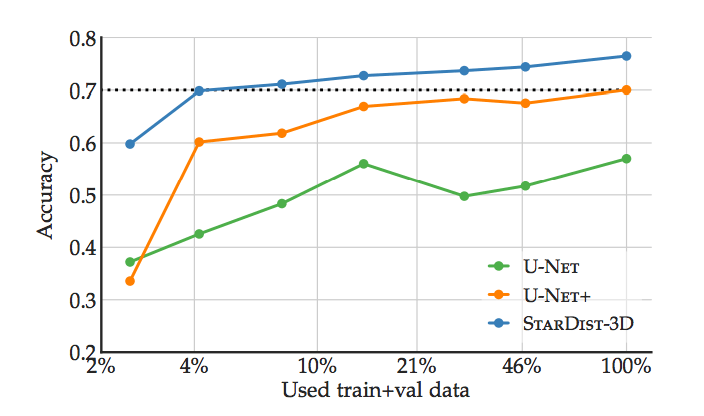
上記は、使用データ量(訓練データ、バリデーションデータ)毎の精度になります。本モデル「STAR-DIST3D」は青線で表されており、既存モデル(U-NET, U-NET+)と比較したとき、高水準かつより安定した精度を発揮していることがわかります。
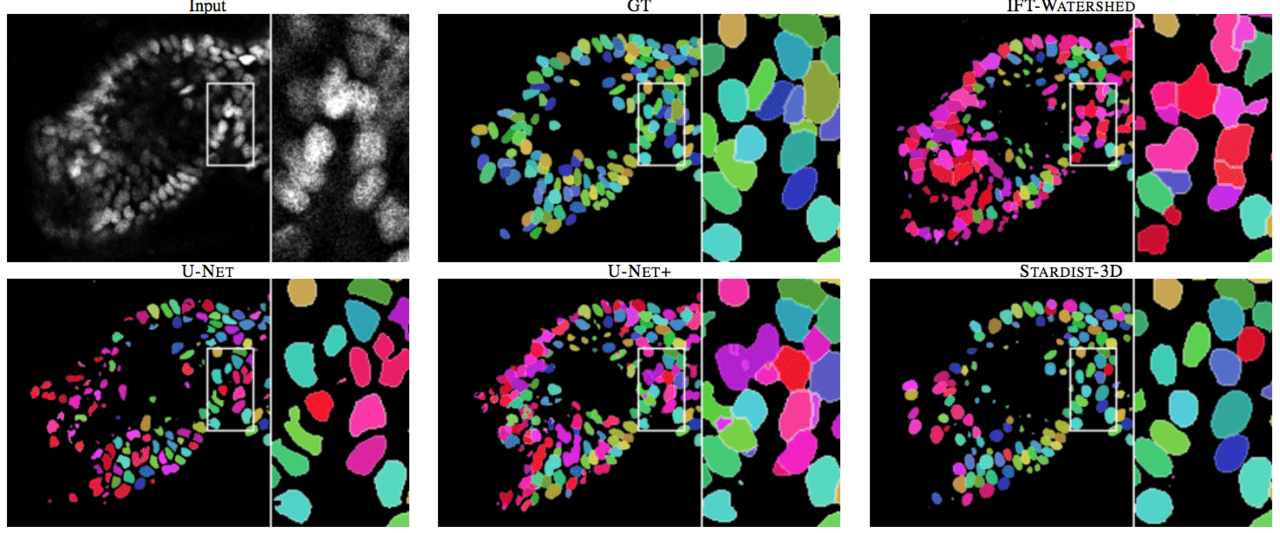
また、上図は既存モデルと「STAR-DIST3D」(右下)の2次元セグメンテーションによる結果を比較したものです。2次元モデルと比較したときも、視覚的にセグメンテーションはよりはっきりと、かつ重複部分が少なく写っていることがわかります。
例えば、既存の最新モデルであるU-NET+(STRADIST-3Dの左)と比較しても、重複部分を含まずセグメンテーションできていることがわかります。
上記(i),(ii)の実験結果から、実際に”星状凸多面体表現”を適用させることで、セグメンテーションがより高精度になることがわかりました。
(ii)3Dモデリング実験

上画像は、「STAR-DIST3D」を用いて、WORM(画像左)とPARYHALE(画像右)の生のデータから3Dモデリングを行なった結果になります。非常に色鮮やかに、かつはっきりとセグメンテーションされたモデリングできているのがわかります。しかし、WORMにおいては、一部分が白くなっており、入力画像から認識されていない部分が残ってしまっているのがわかります。この部分に関しては、モデルを改良してセグメンテーション可能な領域を増やす必要があるでしょう。
終わりに
今回は、”星状凸多面体表現”を用いた細胞核の3D検出・分類を行うモデル「STAR-DIST3D」を紹介しました。既存の2次元セグメンテーションモデルに工夫を加え、異方性をもつデータセットであっても実際に表現できていることがわかりました。
また、「STAR-DIST3D」は既存モデルと比較しても、精度の高いセグメンテーションを行うことができ、星状凸多面体表現による効率的なメカニズムにより、より簡単かつ高速な学習が可能です。
上記の実験結果を踏まえて、「STAR-DIST3D」がセグメンテーションの生成においてより一般的に実用化されるべきであると論文著者は言っています。
星状凸多面体という幾何学的アイデアを、機械学習の計算効率化に導入するのは非常に面白いと思います。理論面において、定義の仕方(計算の仕方)を変更するだけで明確なアップデートを行うことができるのも非常に良い点だと思います。
この記事に関するカテゴリー










